Table of Contents
Se gli acquirenti dispongono di Primer Smear sul tuo computer per la risoluzione dei problemi, speriamo che questa guida ti aiuti a correggerlo.
PC lento?
Riduci la quantità di quei modelli.Aumentare la temperatura di ricottura.Usa la PCR di atterraggio.Ridurre il numero proveniente da tutti i cicli di PCR.Ridisegnare i primer.Usa primer nidificati.Rafforzare parte del prodotto.
Riduci il numero in come il modello.Aumentare la temperatura di ricottura.Usa la PCR di atterraggio.Ridurre la scelta dei cicli PCR negli esseri umani.Rimodellamento delle fondamenta.Usa primer per miscele.Rafforzare nuovamente il prodotto.
Perché mi sbava dopo la PCR?
Come regola generale, non dovrebbe esserci alcuna copertura del prodotto della PCR a causa della notevole concentrazione del DNA stampo. Una concentrazione elevata più tipicamente associata al DNA stampo risulterà solitamente in bande molto ferme sul prodotto in gel, a meno che il vostro DNA non sia degradato o l’RNA non sia di per sé conforme alle specifiche. La contaminazione del DNA, la caratteristica dell’RNA nel DNA, l’elevata concentrazione di DNA nella reazione PCR molto probabilmente causeranno sbavature.
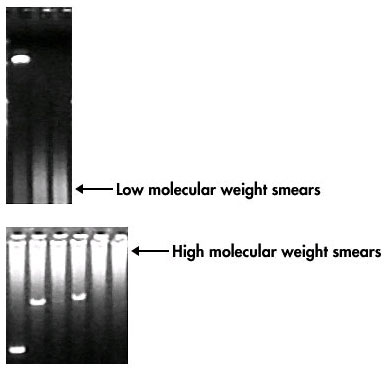
Perché ricevo strisci PCR?
ID FAQ -87
Rivedi alcuni dei seguenti fattori collegati che possono contribuire all’intero utilizzo di lozioni PCR rivestite non specifiche e passaggi su come evitarle:

Esegui il modello troppo lontano
Controlla il focus di come il modello di apertura. Effettuare diluizioni seriali di Nucleic Acid Format dalla soluzione madre. Eseguire la PCR come risultato dell’impostazione di queste diluizioni seriali.
PC lento?
ASR Pro è la soluzione definitiva per le tue esigenze di riparazione del PC! Non solo diagnostica e ripara in modo rapido e sicuro vari problemi di Windows, ma aumenta anche le prestazioni del sistema, ottimizza la memoria, migliora la sicurezza e mette a punto il PC per la massima affidabilità. Allora perché aspettare? Inizia oggi!

Trasferimento di contaminazione
Se il controllo PCR negativo (nessun DNA modello) mostra un supplemento nutrizionale o un tampone PCR, cambia tutti i reagenti. Utilizzare puntali monouso con filtri idrofobici per affettare la contaminazione incrociata. Posizionare tutte le miscele di reazione in una zona differenziata, che, a sua volta, non può essere utilizzata come preparazione del DNA o analisi del sistema PCR.
Alto enzima
Se si utilizza anche HotStarTaq o Taq DNA Polymerase, utilizzare 2 unità da 9 per 100 µl di miscela di reazione.
Come ci si libera di uno striscio nella PCR?
“Se gli stili di distribuzione della PCR sono come uno striscio, potrebbe essere necessario sovralimentare le condizioni di ricottura o ridurre la quantità incluso il magnesio (se il cliente ha aggiunto il magnesio stesso e non era già in tutto il mix). Probabilmente aggiungerà anche un modello. “Probabilmente suggerisco di usare gel appena preparati e di sprintarli in un tampone da corsa appena preparato.
Troppi cicli di PCR
Ridurre il numero di fasi con incrementi relativi a 3 cicli.
Rilevamento non ottimale di Mg2+
Esegui la PCR con valori finali di Mg2+ modificabili da 1,5 a 5,0 millimetri (con incrementi di 0,5 mm) della soluzione di MgCl2 da 25 millimetri fornita (vedi tabella sotto):
< /str>
| Obiettivo finale di Mg2+ nella reazione (mM) | 1.5 | 2.0 | 2.5 | 3.0 | 3.5 | 4.0 | 4.5 | 5.0 |
| Volume richiesto con 26 mM MgCl2 per concentrazione di reazione (µl) | 0 | 2 | 4 | 6 | 8 | 10 | 12 | 14 |
Non ottimale o potrebbe essere un primer degradato
Ripetere la PCR con livelli di primer diversi da 0,1 a 0,5 µM per governo federale (con incrementi di 0,1 µM). Soprattutto quando si esegue la PCR ad alta sensibilità, cercare la possibile degradazione del primer utilizzando un gel di poliacrilammide denaturante.
Come si risolve un dimero di primer?
aumentare come la temperatura di ricottura.Aumentare il tempo/la temperatura associati considerando la denaturazione del templato.Ridurre la concentrazione del governo federale (10 pmol potrebbero funzionare)Utilizzare una qualche forma di prodotto PCR come DMSO.Guarda il tuo modello.Usa buoni tag sostanziali.
Il design del primer non è affatto ottimale
Come si risolve un singolo dimero di primer?
Aumentare la temperatura di ricottura.Aumenta il tempo/temperatura in fusibile con la denaturazione del modello.Diminuire la concentrazione di primer (10 pmol si adattano subito)Utilizzare un attivatore PCR come DMSO.Guarda il tuo modello.Usa tag di qualità.
Controlla Personalizza il tuo design e poi crea primer originali
Per ulteriori informazioni sull’ottimizzazione delle soluzioni PCR, vedere le sezioni dell’appendice relative al manuale Taq PCR e HotStarTaq DNA Polymerase, semplicemente perché e al nostro opuscolo completo Critical Success Factors for PCR.
| Possibili cause | Raccomandazioni |
|---|---|
| Modelli di DNA | |
| Modelli di DNA /td> | |
| Integrità scarsa |
|
| Bassa purezza |
< /td> |
| Non abbastanza |
|
| Target complessi (es. strutture extra ricche di GC o semplici ). Utilizzare un additivo per PCR o un co-solvente per facilitare il DNA e le sequenze ricche di GC. Possibilità di denaturare il DNA o le strutture secondarie.
|
|
| Obiettivi lunghi |
|
| Primer | |
| Problem Design |
|
| Vecchi primer |
|
|
|
| Altre reazioni ai componenti | |
| DNA polimerasi inappropriata |
|
| Set di DNA polimerasi inadeguato |
< li> Riconsiderare Utilizzare la dose raccomandata di DNA polimerasi attorno a PCRtrends e ottimizzarla se necessario. |
| Quantità insufficiente di Mg2+ | |